INTRODUCCIÓN
La segmentación de árboles individuales a partir de datos de Light Detection and Ranging (LiDAR) se ha consolidado como un eje central del monitoreo y la gestión forestal contemporáneos. La delimitación precisa de copas permite derivar atributos estructurales clave que sostienen inventarios, estimaciones de biomasa, evaluaciones ecológicas y decisiones de manejo sostenible (Ferraz et al., 2016; Crowther et al., 2015). Frente a los elevados costos y las restricciones espaciales de los levantamientos de campo, LiDAR ofrece nubes de puntos tridimensionales de alta resolución que habilitan análisis a nivel de árbol, incluso en territorios con relieve complejo y doseles estratificados (Jakubowski et al., 2013; Aubry-Kientz et al., 2019).
Durante las dos últimas décadas se han desarrollado numerosos métodos de segmentación. Los enfoques tradicionales basados en modelos de altura de copa (CHM), como filtros de máximos locales, cuencas de drenaje y crecimiento de regiones, exhiben resultados aceptables en coníferas, pero su desempeño decae en bosques heterogéneos y multilaminares por su sensibilidad a parámetros y su dependencia de proyecciones bidimensionales (Jakubowski et al., 2013). Alternativas como la agrupación por densidad (p. ej., DBSCAN) o los modelos geométricos de directividad de ramas mejoran la delineación en ciertos contextos, aunque siguen condicionadas por la sintonía de parámetros y por subrendimiento en copas densas y entrelazadas (Comesaña-Cebral et al., 2021; Yang et al., 2024).
Los avances recientes en aprendizaje profundo han transformado la investigación al operar directamente sobre nubes de puntos con arquitecturas como PointNet, PointNet++ y detectores basados en CNN, alcanzando mayores F1 y robustez en bosques complejos al aprender rasgos locales y globales de datos no estructurados (Qi et al., 2017; You et al., 2023). No obstante, estos enfoques requieren grandes conjuntos anotados y recursos de cómputo significativos, y aún enfrentan dificultades en copas muy superpuestas, un reto transversal en ITS (Henrich et al., 2024).
Para mitigar estas limitaciones, han surgido métodos híbridos que integran detección de troncos, imágenes espectrales o LiDAR multivista, reduciendo sobre- y subsegmentación y reportando F1 elevados en ambientes frondosos y urbanos (Deng et al., 2024; Yu et al., 2024; Hua et al., 2022). Sin embargo, su complejidad y dependencia de datos multimodales restringen su escalabilidad. En paralelo, los algoritmos de desplazamiento medio adaptativo (AMS3D) ajustan de manera automática el ancho de banda del núcleo según la estructura local del dosel, aumentando la automatización y eficiencia, aunque con limitaciones para detectar el sotobosque (Hu et al., 2017; Chen et al., 2018; Hui et al., 2021).
Una refinación adicional consiste en modelar la forma de la copa mediante relaciones alométricas e integrar núcleos elipsoidales o superelipsoidales que alinean la segmentación con la morfología arbórea, mejorando la exhaustividad y el índice de Jaccard —en especial en coníferas— y produciendo límites de copa más realistas (Xiao et al., 2019; Tusa et al., 2021). Pese a estos avances, gran parte de los estudios se apoyan en datos regionales o propietarios, lo que limita la reproducibilidad y la validación intersitio; a diferencia de otros ámbitos de teledetección donde plataformas abiertas han favorecido ejercicios reproducibles, como el mapeo de NDVI en Ecuador (Álvarez Mendoza et al., 2024). En ITS, los anchos de banda adaptativos y los modelos de copa han sido escasamente evaluados en conjuntos de referencia abiertos.
Por tanto, el objetivo es evaluar el desplazamiento medio adaptativo tridimensional con un modelo de corona para la segmentación de árboles individuales a partir de datos LiDAR obtenidos con vehículos aéreos no tripulados
MÉTODOS
La metodología propuesta operacionaliza el algoritmo de desplazamiento medio adaptativo tridimensional (AMS3D) con modelo de forma de copa para la segmentación de copas individuales de árboles (ITC, por sus siglas en inglés). El diseño integra el preprocesamiento de datos LiDAR, la estimación adaptativa del perfil del núcleo, la calibración de parámetros basada en relaciones alométricas y la evaluación cuantitativa frente a inventarios de referencia. Este flujo de trabajo se fundamenta en los avances recientes en segmentación mediante desplazamiento medio adaptativo (Hu et al., 2017; Yan et al., 2020; Lei et al., 2022) y en modelos sensibles a la forma de la copa (Xiao et al., 2016; Tusa et al., 2021). La Figura 1 resume el flujo metodológico de la presente investigación y se describe a continuación.
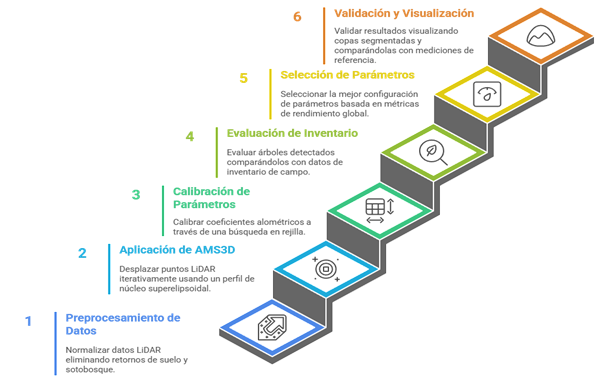
Figura 1. Flujo de trabajo de la metodología AMS3D con modelo de forma de copa.
Elaboración: Los autores.
1. Preprocesamiento de datos LiDAR
Las nubes de puntos LiDAR brutas fueron normalizadas eliminando retornos de suelo y sotobosque por debajo de 1.5 m, siguiendo la práctica común para evitar la influencia de arbustos y vegetación herbácea (Kandare et al., 2016). La elevación del terreno se modeló y se sustrajo, produciendo alturas normalizadas que garantizaron la consistencia entre parcelas de estudio y ofrecieron datos estandarizados para la segmentación.
2. AMS3D con modelo de copa
Cada punto LiDAR ![]() fue desplazado de manera iterativa hacia un
modo local de densidad empleando un perfil de núcleo superelipsoidal (SE) con
exponente
fue desplazado de manera iterativa hacia un
modo local de densidad empleando un perfil de núcleo superelipsoidal (SE) con
exponente ![]() . Esta configuración fue seleccionada porque
ofreció el mejor compromiso entre la exhaustividad (recall) y el índice
de Jaccard en parcelas de coníferas, de acuerdo con lo reportado por Tusa et
al. (2021). Por ello, en este estudio no se realizó comparación con el núcleo
cilíndrico.
. Esta configuración fue seleccionada porque
ofreció el mejor compromiso entre la exhaustividad (recall) y el índice
de Jaccard en parcelas de coníferas, de acuerdo con lo reportado por Tusa et
al. (2021). Por ello, en este estudio no se realizó comparación con el núcleo
cilíndrico.
Los parámetros iniciales del núcleo se definieron como:
![]() [1]
[1]
![]() [2]
[2]
donde ![]() y
y ![]() son coeficientes alométricos, y
son coeficientes alométricos, y ![]() corresponde a la altura normalizada del punto
corresponde a la altura normalizada del punto
![]() . El proceso iterativo calculó el vector de
desplazamiento medio hasta cumplir el criterio de convergencia
. El proceso iterativo calculó el vector de
desplazamiento medio hasta cumplir el criterio de convergencia ![]() . La medida de convergencia
. La medida de convergencia ![]() se definió como la distancia euclidiana entre
estimaciones sucesivas del modo.
se definió como la distancia euclidiana entre
estimaciones sucesivas del modo.
Cuando no se alcanzaba la convergencia, el ancho de banda del núcleo se ajustaba mediante el modelo elipsoidal de copa, estimando los parámetros de la misma:
![]() [3]
[3]
![]() [4]
[4]
donde ![]() es
la altura máxima de los puntos dentro del vecindario cilíndrico de
es
la altura máxima de los puntos dentro del vecindario cilíndrico de ![]() , y
, y ![]() corresponde
al umbral inferior de altura (1.5 m) utilizado para eliminar la vegetación de
sotobosque. El parámetro
corresponde
al umbral inferior de altura (1.5 m) utilizado para eliminar la vegetación de
sotobosque. El parámetro ![]() representa el semieje mayor en dirección
vertical (profundidad de copa), mientras que
representa el semieje mayor en dirección
vertical (profundidad de copa), mientras que ![]() denota el radio estimado de la copa derivado de
la relación alométrica con la altura del árbol.
denota el radio estimado de la copa derivado de
la relación alométrica con la altura del árbol.
El exponente alométrico ![]() se obtuvo igualando el radio del perfil del
núcleo con el radio de la copa a media altura:
se obtuvo igualando el radio del perfil del
núcleo con el radio de la copa a media altura:
![]() [5]
[5]
Finalmente, el radio elipsoidal del núcleo se actualizó mediante:
![]() [6]
[6]
lo cual restringe el tamaño del núcleo de acuerdo con la
geometría del elipsoide ajustado. En este estudio no se aplicó el modelo
híbrido de copa propuesto por Ferraz (2016) y Tusa et al. (2021), que combina
radios cilíndricos y elipsoidales (![]() ), ya que se consideró que el modelo elipsoidal
con
), ya que se consideró que el modelo elipsoidal
con ![]() era suficiente para las condiciones
experimentales.
era suficiente para las condiciones
experimentales.
3. Calibración de parámetros alométricos
Los coeficientes alométricos se calibraron mediante una búsqueda en rejilla sobre intervalos derivados de regresiones lineales entre altura y dimensiones de la copa. Esta calibración empírica exploró múltiples configuraciones por parcela, siguiendo las estrategias adaptativas planteadas por Hu et al. (2017) y Yan et al. (2020).
4. Evaluación frente a inventario de referencia
Los árboles detectados se emparejan con los árboles del inventario de campo utilizando la distancia euclidiana entre las copas. A partir de los pares coincidentes, se calcularon las métricas estándar:
![]() [7]
[7]
![]() [8]
[8]
![]() [9]
[9]
donde ![]() corresponde al número de
verdaderos positivos,
corresponde al número de
verdaderos positivos, ![]() al número de árboles de
referencia y
al número de árboles de
referencia y ![]() al número de detecciones.
al número de detecciones.
5. Selección global de parámetros
Dado que los parámetros óptimos pueden variar entre parcelas, los resultados se agregaron globalmente para identificar la mejor configuración de coeficientes. La selección priorizó maximizar el F1-score global y, en caso de empates, se consideraron criterios adicionales como mayor número de verdaderos positivos, menor número de falsos positivos, mayor Exhaustividad y Precisión.
6. Validación y visualización:
La configuración global seleccionada se
validó mediante la visualización cualitativa de las copas segmentadas y la
comparación cuantitativa con mediciones de referencia. Las detecciones fueron
proyectadas en el plano ![]() , ajustadas a formas
elipsoidales y evaluadas por su plausibilidad ecológica. Esta etapa confirmó la
capacidad de AMS3D con modelo de copa para capturar la variabilidad estructural
en bosques con doseles complejos.
, ajustadas a formas
elipsoidales y evaluadas por su plausibilidad ecológica. Esta etapa confirmó la
capacidad de AMS3D con modelo de copa para capturar la variabilidad estructural
en bosques con doseles complejos.
RESULTADOS
La evaluación experimental de este estudio se realizó utilizando el conjunto de datos abierto publicado por Dubrovin et al. (2024), disponible públicamente en Kaggle (https://www.kaggle.com/datasets/sentinel3734/tree-detection-lidar-rgb/data). Este recurso integra un inventario de campo detallado con nubes de puntos LiDAR adquiridas mediante vehículos aéreos no tripulados (UAV) y ortofotos RGB de alta resolución en bosques mixtos y densos.
El área de estudio
se ubica en Perm Krai, Rusia, aproximadamente a 90 km al este del centro
administrativo regional. El bosque se caracteriza por su estructura mixta y
densa, con un dosel complejo e irregular. El levantamiento de campo comprende
3600 árboles distribuidos en 10 parcelas rectangulares de ![]() m (0.5 ha cada una), con un área total de
5 ha. Todos los árboles están georreferenciados en el sistema de coordenadas
UTM 40N (EPSG:32640). Para cada individuo se registró la especie y el diámetro
a la altura del pecho (DAP), medido con calibradores a 1.3 m del suelo en dos
direcciones perpendiculares y promediado. Aproximadamente el 20 % de los
árboles cuentan con altura medida y el 10 % con edad determinada mediante
núcleos de crecimiento. El inventario incluye siete especies arbóreas,
distribuidas casi equitativamente entre coníferas y caducifolias: abeto,
abedul, abeto blanco, álamo, tilo, aliso y sauce.
m (0.5 ha cada una), con un área total de
5 ha. Todos los árboles están georreferenciados en el sistema de coordenadas
UTM 40N (EPSG:32640). Para cada individuo se registró la especie y el diámetro
a la altura del pecho (DAP), medido con calibradores a 1.3 m del suelo en dos
direcciones perpendiculares y promediado. Aproximadamente el 20 % de los
árboles cuentan con altura medida y el 10 % con edad determinada mediante
núcleos de crecimiento. El inventario incluye siete especies arbóreas,
distribuidas casi equitativamente entre coníferas y caducifolias: abeto,
abedul, abeto blanco, álamo, tilo, aliso y sauce.
El levantamiento LiDAR aéreo se realizó en julio de 2021 bajo condiciones de follaje completo (leaf-on) empleando un sensor AGM-MS3 (frecuencia de adquisición de 640 kHz, alcance de 300 m). El UAV mantuvo una altitud promedio de vuelo de 150 m sobre el terreno, siguiendo el modelo de elevación SRTM. La precisión espacial de las nubes de puntos fue de 3–5 cm. El preprocesamiento incluyó eliminación de duplicados (umbral: 1 mm), filtrado de ruido y clasificación de puntos de terreno, lo que permitió normalizar las alturas. La densidad promedio en las nubes de puntos alcanzó 37 puntos/m².
La Tabla 1 resume las principales características de las 10 parcelas, incluyendo el número de árboles, el tipo de dosel dominante y la densidad promedio de puntos. Este conjunto de datos resulta especialmente adecuado para la evaluación comparativa de algoritmos de segmentación de árboles individuales, al combinar una referencia confiable de campo con información remota multimodal densa.
Tabla 1.
Resumen de las 10 parcelas forestales incluidas en el conjunto de datos.
|
Parcela |
Número de árboles |
Tipo dominante |
Densidad de puntos (pts/m2) |
|
1 |
420 |
Caducifolio |
31.7 |
|
2 |
365 |
Caducifolio |
47.9 |
|
3 |
332 |
Caducifolio |
40.3 |
|
4 |
261 |
Conífero |
33.5 |
|
5 |
208 |
Conífero |
14.2 |
|
6 |
290 |
Conífero |
39.1 |
|
7 |
408 |
Caducifolio |
41.9 |
|
8 |
341 |
Conífero |
35.5 |
|
9 |
490 |
Mixto |
38.2 |
|
10 |
485 |
Mixto |
36.8 |
|
Total |
3600 |
-- |
-- |
Elaboración: Adaptado de Dubrovin et al. (2024).
Este conjunto de datos resulta especialmente adecuado para la evaluación comparativa de algoritmos de segmentación de árboles individuales, ya que combina una referencia confiable obtenida mediante mediciones de campo con datos de teledetección multimodal de alta densidad.
La Tabla 2
sintetiza el rendimiento de segmentación obtenido al explorar una malla de
parámetros de 3x3 para los coeficientes alométricos ![]() y
y ![]() , siguiendo la metodología propuesta por
Tusa et al. (2021). Este diseño experimental respondió a la ausencia de
mediciones de referencia de dimensiones de copa en los datos de campo
disponibles, lo que impidió una calibración directa de las relaciones alométricas.
, siguiendo la metodología propuesta por
Tusa et al. (2021). Este diseño experimental respondió a la ausencia de
mediciones de referencia de dimensiones de copa en los datos de campo
disponibles, lo que impidió una calibración directa de las relaciones alométricas.
Tabla 2.
Resultados globales
de la segmentación de árboles según combinaciones de parámetros (![]() ).
).
|
|
|
|
|
|
|
|
|
|
0.131 |
0.143 |
13757 |
3050 |
10707 |
0.847 |
0.222 |
0.351 |
|
0.131 |
0.786 |
12891 |
2835 |
10056 |
0.787 |
0.22 |
0.344 |
|
0.131 |
0.969 |
12905 |
2795 |
10110 |
0.776 |
0.217 |
0.339 |
|
0.025 |
0.143 |
2889 |
914 |
1975 |
0.254 |
0.316 |
0.282 |
|
0.025 |
0.969 |
2841 |
893 |
1948 |
0.248 |
0.314 |
0.277 |
|
0.025 |
0.786 |
2850 |
885 |
1965 |
0.246 |
0.311 |
0.274 |
|
0.316 |
0.143 |
20558 |
2972 |
17586 |
0.825 |
0.145 |
0.246 |
|
0.316 |
0.969 |
19193 |
2743 |
16450 |
0.762 |
0.143 |
0.241 |
|
0.316 |
0.786 |
19038 |
2703 |
16335 |
0.75 |
0.142 |
0.239 |
Nota: ![]() : Árboles detectados;
: Árboles detectados; ![]() : Verdaderos positivos;
: Verdaderos positivos; ![]() : Falsas detecciones; Exhaustividad:
proporción de árboles de referencia correctamente detectados; Precisión:
proporción de árboles detectados que son correctos; F1-score: media
armónica de Precisión y Exhaustividad;
: Falsas detecciones; Exhaustividad:
proporción de árboles de referencia correctamente detectados; Precisión:
proporción de árboles detectados que son correctos; F1-score: media
armónica de Precisión y Exhaustividad; ![]() .
.
Elaboración: Los autores.
La Tabla 3 detalla los resultados de segmentación obtenidos por parcela, los cuales pueden interpretarse en el contexto de la composición forestal. En términos generales, los resultados revelan contrastes significativos entre rodales caducifolios, coníferos y mixtos, lo que resalta la influencia de la estructura del dosel en el equilibrio entre exhaustividad y precisión.
Tabla 3.
Resultados de segmentación por parcela.
|
|
|
|
|
|
|
1 |
0.657 |
0.381 |
0.483 |
1.454 |
|
2 |
0.86 |
0.22 |
0.35 |
1.212 |
|
3 |
0.861 |
0.205 |
0.332 |
1.235 |
|
4 |
0.939 |
0.151 |
0.261 |
1.12 |
|
5 |
0.99 |
0.115 |
0.207 |
0.895 |
|
6 |
0.89 |
0.191 |
0.314 |
1.238 |
|
7 |
0.85 |
0.273 |
0.413 |
1.279 |
|
8 |
0.93 |
0.212 |
0.345 |
1.091 |
|
9 |
0.791 |
0.3 |
0.435 |
1.249 |
|
10 |
0.846 |
0.297 |
0.439 |
1.164 |
Elaboración: Los autores.
La Figura 2 ilustra el mejor resultado de segmentación, correspondiente a la Parcela 1, que alcanzó el F1 más alto (0.483). Este caso ejemplifica el equilibrio más favorable entre exhaustividad y precisión en todo el conjunto, confirmando que el núcleo AMS3D puede delinear eficazmente las copas dominantes en condiciones favorables, particularmente en rodales caducifolios con densidad de puntos moderada.
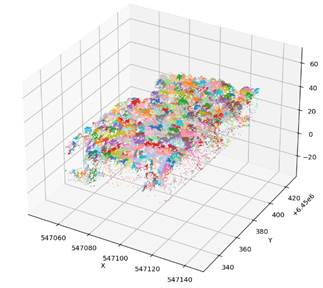
Figura 2. Ejemplo de la mejor parcela segmentada obtenida con la combinación de
parámetros seleccionada (![]() ).
).
Elaboración: Los autores.
DISCUSIÓN
Los resultados de
la Tabla 3 ponen de manifiesto varias fortalezas del enfoque. En primer lugar,
las parametrizaciones con ![]() mostraron valores de exhaustividad
consistentemente altos (0.776–0.847), lo que confirma la robustez del perfil de
núcleo AMS3D para detectar una gran proporción de árboles de referencia. En
particular, la configuración
mostraron valores de exhaustividad
consistentemente altos (0.776–0.847), lo que confirma la robustez del perfil de
núcleo AMS3D para detectar una gran proporción de árboles de referencia. En
particular, la configuración ![]() y
y ![]() logró el mejor equilibrio entre tasa de
detección y exactitud espacial, alcanzando una exhaustividad de 0.847 y un
F1-score de 0.351. Estos hallazgos corroboran estudios previos sobre la
adaptabilidad de los anchos de banda del mean shift a la geometría de
las copas en rodales dominados por coníferas (Hu et al., 2017; Yan et al.,
2020; Lei et al., 2022).
logró el mejor equilibrio entre tasa de
detección y exactitud espacial, alcanzando una exhaustividad de 0.847 y un
F1-score de 0.351. Estos hallazgos corroboran estudios previos sobre la
adaptabilidad de los anchos de banda del mean shift a la geometría de
las copas en rodales dominados por coníferas (Hu et al., 2017; Yan et al.,
2020; Lei et al., 2022).
No obstante,
emergen limitaciones relevantes. La exhaustividad alta se logra a costa
de una baja precisión (≈0.22), reflejando numerosos falsos positivos. Los
anchos de banda pequeños (![]() ) reducen drásticamente la exhaustividad,
mientras que valores mayores (
) reducen drásticamente la exhaustividad,
mientras que valores mayores (![]() ) incrementan los falsos positivos y
disminuyen la precisión por debajo de
) incrementan los falsos positivos y
disminuyen la precisión por debajo de ![]() . Estos resultados subrayan el compromiso
inherente en la segmentación mean shift: maximizar la exhaustividad,
tiende a causar sobresegmentación, mientras que priorizar la precisión reduce
la exhaustividad (Xiao et al., 2019).
. Estos resultados subrayan el compromiso
inherente en la segmentación mean shift: maximizar la exhaustividad,
tiende a causar sobresegmentación, mientras que priorizar la precisión reduce
la exhaustividad (Xiao et al., 2019).
En síntesis, la malla de parámetros ilustra la sensibilidad del algoritmo AMS3D frente a la elección de coeficientes alométricos. Su principal fortaleza radica en la capacidad de detección robusta (exhaustividad alta), pero su limitación es la sobresegmentación sistemática y la baja precisión, especialmente en rodales estructuralmente complejos con copas superpuestas.
Sobre los resultados descritos en la Tabla 3, en las parcelas caducifolias (1, 2, 3 y 7), la exhaustividad varió entre 0.657 y 0.861, mientras que la precisión permaneció modesta (0.205–0.381). La parcela 1 alcanzó el equilibrio más favorable (F1 = 0.483), en contraste con las parcelas 2 y 3, que presentaron precisiones muy bajas pese a altos valores de exhaustividad. Este patrón refleja la irregularidad de las copas y el solapamiento de ramas en especies de hoja ancha, que incrementan las detecciones falsas. En la parcela 7, con la mayor densidad de árboles dentro de las caducifolias, se alcanzó un F1 de 0.413, lo que sugiere que la mayor densidad de copas proporciona máximos locales más consistentes, aunque con cierta sobresegmentación.
Los rodales de coníferas (parcelas 4, 5, 6 y 8) exhibieron los valores más altos de exhaustividad, destacando la parcela 5 con 0.990. Sin embargo, ello se acompañó de una precisión muy baja (0.115) y el menor F1-score del conjunto (0.207). Esta marcada asimetría indica que, aunque las copas dominantes fueron detectadas de manera confiable, el algoritmo tendió a sobresegmentar copas cónicas bajo parámetros de núcleo fijos. Las parcelas 4, 6 y 8 mostraron un comportamiento similar: exhaustividad elevada (0.890–0.939) pero precisión menor al 0.21.
En los rodales mixtos (parcelas 9 y 10), el algoritmo alcanzó los resultados más equilibrados, con F1-scores de 0.435–0.439 y precisiones cercanas al 0.30, manteniendo exhaustividad entre 0.791 y 0.846. La heterogeneidad de especies parece mitigar sesgos sistemáticos: las copas caducifolias reducen la fragmentación excesiva de coníferas, mientras que las copas de coníferas incrementan la completitud, produciendo un compromiso más estable entre errores de omisión y comisión.
En cuanto a la exactitud posicional, los errores medios en distancia XY oscilaron entre 0.895 m y 1.454 m. Las parcelas caducifolias presentaron desviaciones mayores, como en el caso de la parcela 1 (1.454 m), asociadas a la geometría irregular de las copas. Por el contrario, las parcelas de coníferas, particularmente la parcela 5 (0.895 m), registraron menores errores debido a la simetría de sus copas. Los rodales mixtos se situaron en un rango intermedio (1.164–1.249 m).
En síntesis, los rodales caducifolios evidencian los desafíos que plantean las copas irregulares, los coníferos logran elevados niveles de detección con baja precisión, y los rodales mixtos ofrecen el mejor compromiso. Estos hallazgos confirman que la composición forestal influye directamente en el desempeño de la segmentación y refuerzan la necesidad de modelos adaptativos de forma de copa para reducir falsas detecciones en rodales homogéneos (Hu et al., 2017; Tusa et al., 2021; Yan et al., 2020).
CONCLUSIONES
Este estudio evaluó el algoritmo Adaptive Mean Shift 3D (AMS3D) con modelado de forma de copa para la segmentación de árboles individuales a partir de datos LiDAR. Los experimentos demostraron que el método fue altamente efectivo en la detección de árboles de referencia, con valores de recall consistentemente superiores a 0.75 en la mayoría de las parametrizaciones. Esta fortaleza fue particularmente evidente en rodales de coníferas y mixtos, donde las copas simétricas o heterogéneas facilitaron una identificación robusta de los ápices. No obstante, la precisión se mantuvo baja en todas las parcelas debido a la sobresegmentación sistemática, especialmente en rodales homogéneos de coníferas donde el algoritmo tendió a dividir en exceso las copas. Las parcelas caducifolias evidenciaron la dificultad adicional de las geometrías irregulares y las copas superpuestas, mientras que los rodales mixtos alcanzaron el equilibrio más estable entre errores de omisión y comisión.
Estos hallazgos confirman el potencial del AMS3D con
núcleos informados, a la vez que subrayan su sensibilidad frente a la elección
de los parámetros alométricos. Futuros trabajos deberían explorar estrategias
para automatizar la estimación de ![]() y
y ![]() , mediante
segmentaciones bidimensionales preliminares de modelos de altura de copa,
reduciendo así el ajuste manual. Otra dirección es la integración de datos
multimodales, en particular la información RGB disponible en el conjunto de
datos, con el fin de mejorar la delimitación de los límites de copa.
Finalmente, se recomienda realizar estudios comparativos con enfoques de
aprendizaje profundo, dado que estos métodos pueden aprovechar información
estructural más rica, aunque requieren delineaciones de copa más coherentes.
Tales extensiones contribuirán a potenciar el uso de AMS3D en inventarios
forestales y aplicaciones ecológicas.
, mediante
segmentaciones bidimensionales preliminares de modelos de altura de copa,
reduciendo así el ajuste manual. Otra dirección es la integración de datos
multimodales, en particular la información RGB disponible en el conjunto de
datos, con el fin de mejorar la delimitación de los límites de copa.
Finalmente, se recomienda realizar estudios comparativos con enfoques de
aprendizaje profundo, dado que estos métodos pueden aprovechar información
estructural más rica, aunque requieren delineaciones de copa más coherentes.
Tales extensiones contribuirán a potenciar el uso de AMS3D en inventarios
forestales y aplicaciones ecológicas.
FINANCIAMIENTO
No monetario.
AGRADECIMIENTO
Expreso mi más sincero agradecimiento al Programa de Maestría en Matemática Aplicada, Mención Matemática Computacional, de la Universidad Nacional de Chimborazo, por brindarme la oportunidad de formarme académicamente y desarrollar esta investigación en un entorno de excelencia académica y científica.
REFERENCIAS CONSULTADAS
Álvarez Mendoza, C. I., Mollocana, J. G., y Gualotuna, D. (2024). Mapping of NDVI in Ecuador during the last 20 years using the Google Earth Engine cloud geospatial tool. En M. V. Garcia, C. Gordón-Gallegos, A. Salazar-Ramírez & C. Nuñez (Eds.), Proceedings of the International Conference on Computer Science, Electronics and Industrial Engineering (CSEI 2023). Springer Nature. https://doi.org/10.1007/978-3-031-70981-4_1
Aubry Kientz, M., Dutrieux, R., Ferraz, A., Saatchi, S., Hamraz, H., Williams, J., Coomes, D., Piboule, A., y Vincent, G. (2019). A comparative assessment of the performance of individual tree crowns delineation algorithms from ALS data in tropical forests. Remote Sensing, 11(9), 1086. https://doi.org/10.3390/rs11091086
Chen, W., Hu, X., Chen, W., Hong, Y., y Yang, M. (2018). Airborne LiDAR remote sensing for individual tree forest inventory using trunk detection-aided mean shift clustering techniques. Remote Sensing, 10(7), 1078. https://doi.org/10.3390/rs10071078
Comesaña Cebral, L., Martínez Sánchez, J., Lorenzo, H., & Arias, P. (2021). Individual tree segmentation method based on mobile backpack LiDAR point clouds. Sensors, 21(18), 6007. https://doi.org/10.3390/s21186007
Crowther, T. W., Glick, H. B., Covey, K. R., Bettigole, C., Maynard, D. S., Thomas, S. M., Bradford, M. A. (2015). Mapping tree density at a global scale. Nature, 525(7568), 201–205. https://doi.org/10.1038/nature14967
Deng, S., Jing, S., & Zhao, H. (2024). A hybrid method for individual tree detection in broadleaf forests based on UAV-LiDAR data and multistage 3D structure analysis. Forests, 15(6), 1043. https://doi.org/10.3390/f15061043
Dubrovin, I., Fortin, C., & Kedrov, A. (2024). An open dataset for individual tree detection in UAV LiDAR point clouds and RGB orthophotos in dense mixed forests. Scientific Reports, 14(1), 72669. https://doi.org/10.1038/s41598-024-72669-5
Ferraz, A., Saatchi, S., Mallet, C., & Meyer, V. (2016). Lidar detection of individual tree size in tropical forests. Remote Sensing of Environment, 183, 318–333. https://doi.org/10.1016/j.rse.2016.05.028
Henrich, J., van Delden, J., Seidel, D., Kneib, T., & Ecker, A. S. (2024). TreeLearn: A deep learning method for segmenting individual trees from ground-based LiDAR forest point clouds. Ecological Informatics, 84, 102888. https://doi.org/10.1016/j.ecoinf.2024.102888
Hua, Z., Xu, S., & Liu, Y. (2022). Individual tree segmentation from side-view LiDAR point clouds of street trees using Shadow-Cut. Remote Sensing, 14(22), 5742. https://doi.org/10.3390/rs14225742
Hu, X., Chen, W., & Xu, W. (2017). Adaptive mean shift-based identification of individual trees using airborne LiDAR data. Remote Sensing, 9(2), 148. https://doi.org/10.3390/rs9020148
Hui, Z., Li, N., Xia, Y., Cheng, P., & He, Y. (2021). Individual tree extraction from UAV LiDAR point clouds based on self-adaptive mean shift segmentation. ISPRS Annals of the Photogrammetry, Remote Sensing and Spatial Information Sciences, V(1), 25–30. https://doi.org/10.5194/isprs-annals-V-1-2021-25-2021
Jakubowski, M. K., Li, W., Guo, Q., & Kelly, M. (2013). Delineating individual trees from lidar data: A comparison of vector- and raster-based segmentation approaches. Remote Sensing, 5(9), 4163–4186. https://doi.org/10.3390/rs5094163
Kandare, K., Ørka, H. O., Chan, J. C. W., & Dalponte, M. (2016). Effects of forest structure and airborne laser scanning point cloud density on 3D delineation of individual tree crowns. European Journal of Remote Sensing, 49(1), 337–359. https://doi.org/10.5721/EuJRS20164919
Lei, L., Yin, T., Chai, G., Li, Y., Wang, Y., Jia, X., & Zhang, X. (2022). A novel algorithm of individual tree crowns segmentation considering three-dimensional canopy attributes using UAV oblique photos. International Journal of Applied Earth Observation and Geoinformation, 112, 102893. https://doi.org/10.1016/j.jag.2022.102893
Qi, C. R., Su, H., Mo, K., & Guibas, L. J. (2017). PointNet: Deep learning on point sets for 3D classification and segmentation. En Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition (CVPR) (pp. 77–85). IEEE. https://doi.org/10.1109/CVPR.2017.16
Tusa, E., Monnet, J. M., Barre, J. B., Dalla Mura, M., Dalponte, M., & Chanussot, J. (2021). Individual tree segmentation based on mean shift and crown shape model for temperate forest. IEEE Geoscience and Remote Sensing Letters, 18(12), 2052–2056. https://doi.org/10.1109/LGRS.2020.3012718
Xiao, W., Zaforemska, A., Smigaj, M., Wang, Y., & Gaulton, R. (2019). Mean shift segmentation assessment for individual forest tree delineation from airborne LiDAR data. Remote Sensing, 11(11), 1263. https://doi.org/10.3390/rs11111263
Xiao, W., Xu, S., Oude Elberink, S., & Vosselman, G. (2016). Individual tree crown modeling and change detection from airborne LiDAR data. IEEE Journal of Selected Topics in Applied Earth Observations and Remote Sensing, 9(8), 3467–3477. https://doi.org/10.1109/JSTARS.2016.2541780
Yang, Z., Su, Y., Li, W., Cheng, K., Guan, H., Ren, Y., Hu, T., Xu, G., & Guo, Q. (2024). Segmenting individual trees from terrestrial LiDAR data using tree branch directivity. IEEE Journal of Selected Topics in Applied Earth Observations and Remote Sensing, 17, 956–969. https://doi.org/10.1109/JSTARS.2023.3334014
Yan, W., Guan, H., Cao, L., Yu, Y., Li, C., & Lu, J. Y. (2020). A self-adaptive mean shift tree-segmentation method using UAV LiDAR data. Remote Sensing, 12(3), 515. https://doi.org/10.3390/rs12030515
You, H., Liu, Y., Lei, P., Qin, Z., & You, Q. (2023). Segmentation of individual mangrove trees using UAV-based LiDAR data. Ecological Informatics, 77, 102200. https://doi.org/10.1016/j.ecoinf.2023.102200
Yu, J., Lei, L., & Li, Z. (2024). Individual tree segmentation based on seed points detected by an adaptive crown shaped algorithm using UAV-LiDAR data. Remote Sensing, 16(5), 825. https://doi.org/10.3390/rs16050825
Zhu, L. (2024). GeoSep-PointNet++: A 3D point cloud semantic segmentation algorithm based on geometric information enhancement and feature separation encoding. En Proceedings of the 2024 IEEE 4th International Conference on Information Technology, Big Data and Artificial Intelligence (ICIBA) (pp. 1514–1519). IEEE. https://doi.org/10.1109/ICIBA62489.2024.10869004
©2025 por los autores. Este artículo es de acceso abierto y distribuido según los términos y condiciones de la licencia Creative Commons Atribución-NoComercial-CompartirIgual 4.0 Internacional (CC BY-NC-SA 4.0) (https://creativecommons.org/licenses/by-nc-sa/4.0/).